
塑料污染已成为全球性环境危机。面对每年产生的数亿吨塑料垃圾,科学家们将目光投向自然界的“食塑生物”。近期,北京理工大学杨宇团队在《Applied and Environmental Microbiology》发表研究,从黄粉虫肠道中发现一种特殊酵母菌,其分泌的两种新型角质酶SiCut1和SiCut2,具有塑料废弃物解聚和回收的潜力。它们的氨基酸序列与所有先前描述的角质酶的相同性低于25%,具有独特GYSKG基序的保守S-D-H催化三联体。
01
塑料危机:我们为何需要生物降解技术?
全球每分钟就有一卡车塑料被倒入海洋,传统填埋、焚烧不仅产生二噁英等剧毒物质,更让石油基塑料的碳足迹雪上加霜。机械回收虽能部分缓解,但反复加工导致材料性能断崖式下降,最终难逃填埋命运。酶法回收技术为塑料回收提供了创新方向:利用微生物产生的酶在温和条件下将塑料分解为可循环利用的单体分子,这种工艺不仅降低了传统回收过程中的能耗需求,也为材料的多次循环再生提供了可能。

(图片来源于网络)
02
虫虫特工队:黄粉虫肠道藏着塑料分解大师
研究从黄粉虫肠道中分离出五种酵母,通过固体培养基实验发现,只有BIT-D3能在添加柠檬酸盐、丙酮酸盐或琥珀酸盐作为额外碳源时,在含聚己内酯(PCL)的培养基上形成透明区,表明其具备降解聚酯的能力,而在葡萄糖、乳糖或无额外碳源条件下则无此效果。进一步实验显示,BIT-D3的无细胞上清液能在液体培养基中显著降解PCL,48小时后乳液几乎完全澄清,光密度(OD600)大幅下降,且傅里叶变换红外光谱(FTIR)证实酯键断裂,证明聚酯被分解。
菌株BIT-D3在YPD琼脂上形成橙红色、圆形平滑菌落,直径1.0-1.5毫米,电镜观察显示其为短杆状细胞(5-7 µm × 3-4 µm)。它可在10-40℃和pH 5.0-9.0条件下生长,最适条件为30℃和pH 7.0。基因序列分析表明,BIT-D3与Sakaguchia属的Sakaguchia dacryoidea相似度为92.11%,多基因分析进一步确认其与Sakaguchia cladiensis亲缘关系密切,因此被归类为Sakaguchia属,命名为Sakaguchia sp. BIT-D3。这一发现展示了BIT-D3在聚酯降解中的潜力,为塑料生物降解研究提供了新视角。

图1 黄粉虫肠道酵母的发现与鉴定。(a)BIT-D3在含PCL的培养基上形成透明水解圈。(b)粗酶液降解PCL的实时吸光度变化。(c)FTIR光谱显示酯键特征峰消失。(d)BIT-D3菌落形态。(e)扫描电镜下的短杆状酵母细胞。(f)BIT-D3的系统发育树定位。
03
基因猎手:从12,593个蛋白中锁定“塑料杀手”
由于聚酯塑料分子量过高无法直接被微生物吸收,降解需通过胞外酶实现。研究通过基因组和转录组分析探索了BIT-D3分泌的聚酯降解酶。BIT-D3全基因组测序预测出12,593种蛋白,其中通过SignalP-6.0等工具分析,筛选出84种经典分泌蛋白和5,429种非经典分泌蛋白,总计5,513种分泌蛋白。
为识别聚酯降解相关酶,研究比较了BIT-D3在含柠檬酸盐和葡萄糖的YES液体培养基中的转录组,结果显示,在柠檬酸盐条件下,908种分泌蛋白显著上调。通过UniProt数据库比对,其中两蛋白(A02567和A02569)与Rhizoctonia solani的潜在角质酶相似度最高(46.9%和46.2%),被标注为“类角质酶”,命名为SiCut1和SiCut2。这两种酶可能负责断裂聚酯酯键.系统发育树表明它们与酵母角质酶聚类但形成独立分支。

图2 角质酶的基因组挖掘与进化分析。(a)分泌蛋白筛选流程(信号肽→跨膜域→亚细胞定位→GPI锚定)。(b)柠檬酸盐诱导的分泌蛋白差异表达热图。(c)角质酶系统发育树(新型酶形成独立分支)。
序列分析显示,SiCut1和SiCut2与其他已知角质酶相似度较低(<24.2%),但含保守的催化三联体(S91, D145, H157)和独特的GYSKG基序。AlphaFold2预测的3D结构由五条β折叠和四条α螺旋组成,与已知酵母角质酶CLE(六条β折叠)结构相近,均具狭窄沟槽、无盖结构。电势和疏水性分析显示其独特性。这些特性提示SiCut1和SiCut2可能是新型酵母来源的角质酶。

图3 两种新型角质酶的序列与结构特征
04
塑料克星的秘密武器:SiCut1/SiCut2性能初探
研究者在毕赤酵母(P. pastoris GS115)中利用AOX1启动子过表达这两种酶。SDS-PAGE分析显示,培养基上清液中主要检测到18.2 kDa的蛋白,伴随少量其他分泌蛋白。在7天培养期间,SiCut1和SiCut2的蛋白浓度逐渐上升,分别达到峰值160±2 µg/mL和108±7 µg/mL。以pNP-丁酸酯(pNPB)为底物测定酶活,结果显示两者活性逐渐增加,分别达到0.41±0.03 U/mL和0.37±0.02 U/mL。
酶的最适pH分别为SiCut1为7.0,SiCut2为8.0,且两者在pH 7.0时稳定性最高,表明它们在中性条件下更活跃。温度方面,SiCut1的最适温度为35℃,而SiCut2为45℃。在30℃以下,SiCut1的热稳定性优于SiCut2;但在30℃以上,SiCut2表现出更强的热稳定性。半衰期分析进一步显示,SiCut1在30℃以下更稳定,而SiCut2在30℃以上显著优于SiCut1。这表明SiCut2在较高温度下更具耐热性。这些特性揭示了SiCut1和SiCut2在不同环境条件下的潜在应用价值,为聚酯生物降解研究提供了新的候选酶。
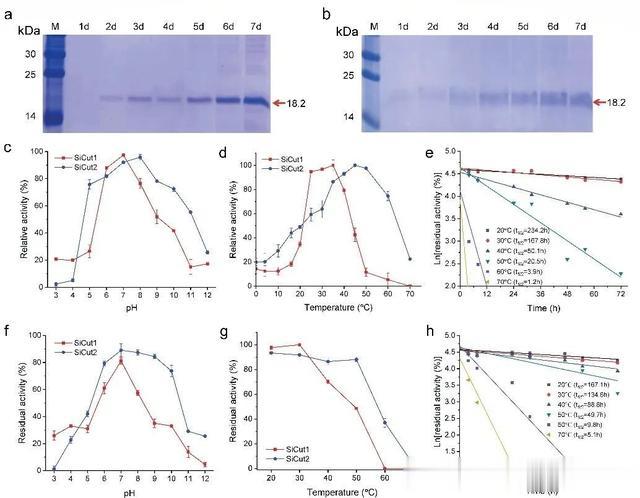
图4 两种新型角质酶的序列与结构特征。(a-b) 毕赤酵母表达SDS-PAGE(第7天产量达160μg/mL)。(c-d) 酶活最适pH与温度曲线。(e-h) 热稳定性与半衰期对比(SiCut2高温更稳定)。
05
实战检验:这些酶能分解哪些塑料?
研究评估了SiCut1和SiCut2在降解多种合成聚酯塑料(如PCL薄膜、PBS薄膜、聚酯-PUR泡沫、PBAT薄膜、PET薄膜、PLA薄膜和聚酯-PUR薄膜)中的能力,实验通过48小时的酶解反应进行。结果显示,SiCut1对PCL薄膜降解效果显著,重量损失达98.0%±2.2%,PBS薄膜和聚酯-PUR泡沫也出现明显碎裂,重量损失分别为12.1%±1.0%和22.2%±1.4%,但对PBAT、PET、PLA和PUR薄膜几乎无作用。SiCut2则对PCL薄膜和聚酯-PUR泡沫有一定降解能力,重量损失分别为21.4%±3.3%和4.3%±1.1%,但对其他材料影响甚微。
通过高效液相色谱-质谱(HPLC-MS)分析降解产物,发现SiCut1和SiCut2降解PCL薄膜产生单体(如6-羟基己酸)和二聚体,表明酯键断裂。SiCut1降解PBS薄膜生成二聚体和三聚体,而聚酯-PUR泡沫在两种酶作用下释放出多种聚酯片段,表明其聚醇结构被分解。PBAT、PET、PLA和聚酯-PUR薄膜未检测到降解产物,与重量变化一致。这些结果显示SiCut1在降解PCL、PBS和聚酯-PUR方面更有效,而SiCut2作用较为有限,为聚酯塑料生物降解提供了重要参考。

图5 (a) 塑料薄膜处理前后形态对比(PCL近乎完全消失)。(b) 不同塑料重量损失率柱状图。(c-e) PCL/PBS/聚氨酯降解产物质谱鉴定。(f-i) PET等材料无产物检出。
06
微观解密:结构决定功能!
研究通过分子对接分析了SiCut1和SiCut2与PCL三聚体(3PCL)的结合模式,发现两者的催化三联体通过亲核攻击3PCL的羰基碳原子实现催化,氧负离子孔形成氢键稳定底物。尽管两者序列同源性高达74.2%,SiCut1对PCL的降解活性显著高于SiCut2。为探究原因,研究通过分子动力学模拟发现,SiCut1的催化位点丝氨酸(S91)与底物断裂位点的平均距离(D1)短于SiCut2,且SiCut1与3PCL的结合自由能比SiCut2低6.57 kJ/mol,表明SiCut1与底物结合更紧密,形成共价中间体的能力更强,从而解释了其更高的PCL降解活性。
此外,SiCut2在30℃以上表现出比SiCut1更优的热稳定性。研究通过B因子和均方根波动(RMSF)分析蛋白质结构刚性,发现SiCut2的环区(G121-V126)相对B因子低于SiCut1对应区域(G122-T127),表明其结构更刚性。RMSF结果进一步显示,在50℃下SiCut2该环区的波动显著小于SiCut1,印证了其更高的热稳定性来源于结构刚性的增强。
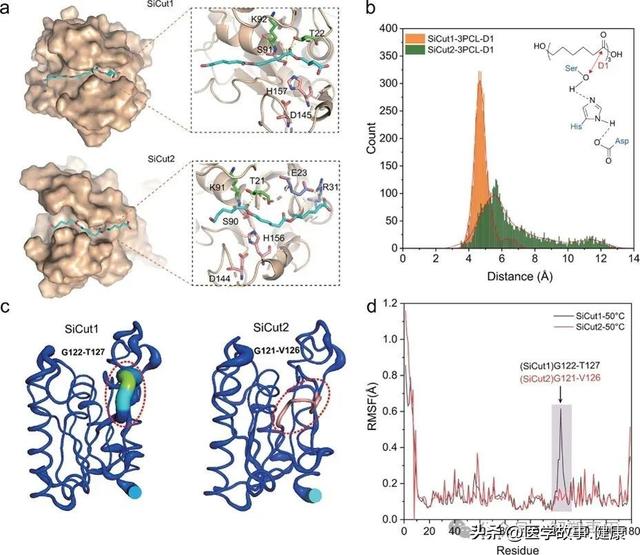
图6 (a) PCL三聚体与酶活性中心的对接模型。(b) 催化丝氨酸与底物距离的动态分布。(c) 蛋白质B因子对比(蓝色表示高刚性区域)。(d) 分子动力学模拟的原子波动分析。
07
结语
这项研究不仅揭示了昆虫肠道微生物在塑料降解中的新机制,也为生物催化回收技术提供了高性能酶资源。未来,随着酶工程技术的进步,这类“自然馈赠”的催化剂有望成为解决塑料污染的关键工具!
